DNA编码的荧光成像技术照亮了细胞核心成分
发布时间:2023-06-14 08:00:00 阅读数: 98
由于细胞的许多核心成分--如DNA、RNA、蛋白质和脂质--只有几纳米大小,大大小于传统光镜的分辨率限制,因此这些分子和结构的确切组成和排列往往是未知的。这导致对生物学的基本方面缺乏机械性的了解。
马克斯-普朗克生物化学研究所和慕尼黑路德维希-马克西米利安大学的研究人员利用最近对超分辨率成像(包括单分子定位显微镜或SMLM)的改进,开发了一种技术,将荧光显微镜的分辨率提高到了埃级水平。研究人员的技术使得对整个完整细胞的研究超过了单个蛋白质--一直到DNA中两个相邻碱基之间的距离。
来自Ralf Jungmann研究小组的研究人员将这项技术称为 "通过连续成像提高分辨率",或称 "RESI"。
目前的SMLM通过在时间上分离结构的单个荧光发射来解决10纳米量级的结构。由于单个目标在一个黑暗的视野中随机发光,它们的位置可以被确定为亚衍射精度。用于纳米级拓扑成像的DNA点积累的SMLM技术,或称DNA-PAINT,使用染料标记的DNA "成像器 "链与它们的目标结合的互补物的瞬时杂交来实现实现超分辨率所需的发光。
然而,到目前为止,无论是DNA-PAINT还是其他超分辨率方法都无法解决最小的细胞结构。
RESI建立在DNA-PAINT的基础上,利用其通过DNA序列编码目标身份的能力。通过用不同的DNA链标记相邻的目标(彼此太近,甚至无法用超分辨率显微镜分辨),在样品中引入了额外的区分度--条形码。通过依次对一个和另一个序列进行成像,从而捕获完整的目标,现在可以明确地分离这些链。
重要的是,由于它们是按顺序成像的,目标可以任意地接近彼此,这是现有技术无法解决的动态。此外,RESI不需要专门的仪器,可以用任何标准的荧光显微镜来应用。
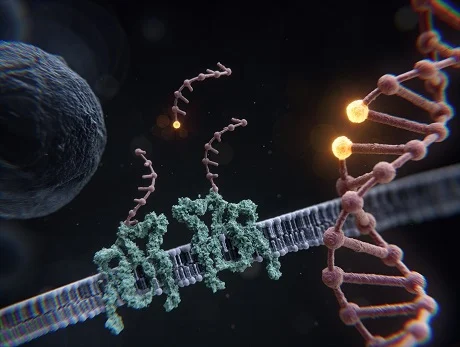
RESI能够以埃的分辨率进行跨长度尺度的显微观察:从整个细胞到单个蛋白质,直至DNA中两个相邻碱基之间的距离。Max Iglesias/Max Planck生物化学研究所提供。
为了证明RESI与其他方法相比在分辨率上的飞跃,研究人员试图解决沿着DNA双螺旋的单个碱基之间的分离问题,这些碱基之间的分离不到1纳米。他们设计了一个DNA折纸纳米结构,呈现出从双螺旋中以一个碱基对的距离突出的单链DNA序列。
研究人员随后对这些单链进行了连续成像,并解决了相邻碱基之间0.85纳米,或8.5埃的距离。他们完成了这些测量,精度为1埃,或百亿分之一米。
据研究人员称,该技术是通用的,其应用范围超出了DNA纳米结构。在单独的测试中,Jungmann和他的团队调查了利妥昔单抗的分子作用方式,这是一种用于治疗CD20阳性血癌的抗CD20单克隆抗体。调查这种药物分子对分子受体模式的影响已经超出了传统显微镜技术的空间分辨率能力。了解这种模式在健康和疾病中以及在治疗时是否以及如何变化,对于机理研究和靶向治疗的设计非常重要。
研究人员利用RESI揭示了未经治疗的细胞中CD20受体作为二聚体的自然排列,并揭示了CD20如何在药物治疗时重新排列成二聚体链。
由于RESI是在完整的细胞中进行的,该技术缩小了X射线晶体学或低温电子显微镜等纯结构技术与传统的低分辨率全细胞成像方法之间的差距。
该研究发表在《Nature》(www.doi.org/10.1038/s41586-023-05925-9)上。
本文由光电查搜集整理,未经同行评议,请自行判断可信度。仅供学习使用。